Indice
Abstract: Proteins are key biological macromolecules which play a pivotal role both in living organisms and in chemical and pharmaceutical research. One of the major obstacles in understanding these polymers has long been the experimental determination of their tridimensional structures. This field of research has recently drawn the attention of the major Artificial Intelligence (AI) companies which have developed highly sophisticated algorithms aimed at solving this scientific problem. This article underlines the fact that these AI programs not only have been able to predict the 3-D structures of every known protein found in nature, in the last two years, but also to create artificial proteins from scratch. As a consequence, this will greatly speed up research ranging from designing new drugs to tackling technological and energetic challenges.
Keywords: struttura tridimensionale delle proteine; Intelligenza Artificiale; algoritmi di deep learning; proteine mutate; progettazione di proteine artificiali; proteine intrinsecamente disordinate
la ricerca in campo chimico, biologico e farmaceutico
Il mio interesse per le proteine e la loro struttura spaziale è nato durante gli anni del dottorato di ricerca, quando queste importanti molecole biologiche rappresentavano dei bersagli sui quali modellare potenziali candidati farmaci. Poi sono sopraggiunti la mancanza di fondi e un esplicito invito da parte dei docenti universitari ad allontanarmi da questo fantastico universo. L’invito consisteva, infatti, nell’investire risorse nel Superenalotto, se avessi voluto trovare un lavoro davvero remunerativo.
L’inaspettato ritorno di fiamma verso questo mondo ha coinciso con la recente impresa di alcuni giganti dell’hi-tech che attraverso lo sviluppo di software di Intelligenza Artificiale (tra i quali AlphaFold, rilasciato da DeepMind, ProteinMPNN e Meta AI) sono riusciti a delucidare la struttura di quasi tutte le proteine conosciute, e non solo. Questi programmi stanno addirittura raggiungendo ottimi risultati nella progettazione e nel design di nuove proteine [1-5].
Se non avessi avuto l’esperienza di cui parlavo, mi sarei forse chiesto il perché dell’interesse di questi colossi tecnologici a investire risorse e dedicare del tempo prezioso ad un argomento ritenuto specialistico e quasi “da intenditori”, quando potrebbero esserci altri settori scientifici, ritenuti ben più stimolanti, da indagare.
Ebbene, occorre innanzitutto chiarire che indagare l’universo delle proteine è molto più che un mero esercizio accademico.
Le proteine, ad esempio, contengono già nel nome uno dei motivi della loro importanza negli organismi viventi, derivando dal termine greco proteios che, un po’ immodestamente, le mette “al primo posto”. Ma se questo non bastasse, potremmo aggiungere che le proteine sono coinvolte in fondamentali processi biologici; che l’emoglobina è una proteina che assicura il trasporto dell’ossigeno nel sangue, e che sono delle proteine a permettere la contrazione muscolare e talvolta a fare la differenza tra un oro olimpico e un rimpianto mondiale. Sono inoltre degli integratori a base di proteine a riempire i pensieri degli amanti del fitness e anche quando chiediamo al parrucchiere di fiducia di eseguire una permanente andiamo a modificare la struttura di specifiche proteine.
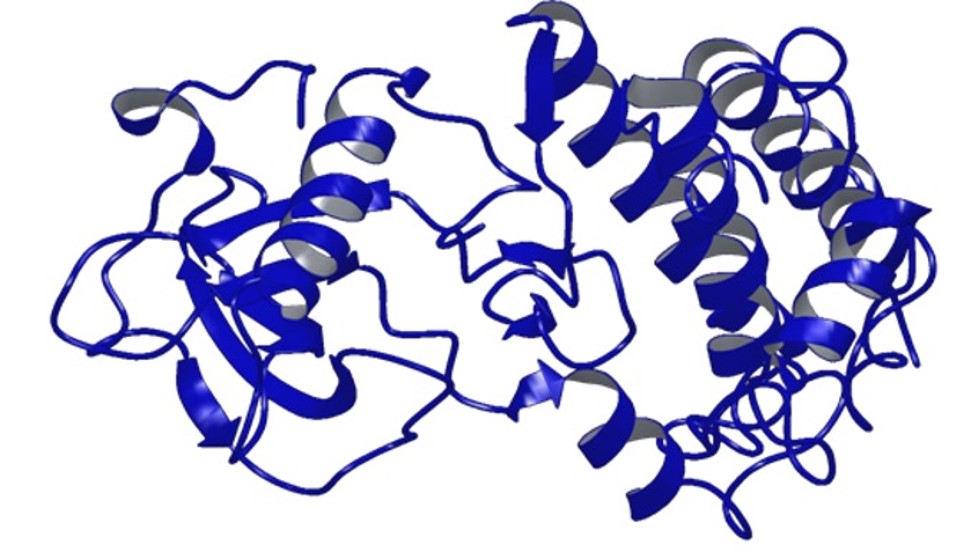 |
Modello di proteina umana – codice 2OH4 (Fonte: Protein Data Bank (PDB) - https://www.rcsb.org)
Ciò, tuttavia, non sarebbe sufficiente a giustificare l’interesse degli scienziati e delle aziende hi-tech verso queste straordinarie molecole biologiche. Dietro la conoscenza della forma tridimensionale ordinata che caratterizza la gran parte delle proteine naturali si cela, infatti, la chiave per lo sviluppo di nuovi farmaci, la comprensione dell’origine di numerose patologie umane e la messa a punto di tecnologie da impiegare nelle sfide epocali del cambiamento climatico e dell’inquina- mento.
Come l’universo in cui viviamo è formato soltanto da una novantina di elementi chimici naturali, tutta la diversità delle forme proteiche si origina da appena una ventina di amminoacidi. Queste piccole unità hanno generalmente dei nomi esotici, dal triptofano alla glicina, per i quali a prima vista è difficile comprendere la derivazione etimologica, all’asparagina, che deve il suo nome a una prelibatezza culinaria, o almeno così la ritengono alcuni.
Si potrebbero immaginare le strutture proteiche, formate dalla successione di centinaia, talvolta migliaia, di questi amminoacidi, come dei nastri da ginnastica ritmica fatti roteare nello spazio da leggiadre ginnaste che vanno ad assumere miriadi di forme diverse.
Il fantastico mondo delle proteine da un po’ di tempo ha ricevuto un interesse sempre crescente da parte di algoritmi di Intelligenza Artificiale. Questi software, in grado di imitare le capacità dell’intelligenza umana, erano in precedenza associati prevalentemente a programmi per le traduzioni di testi e il riconoscimento delle immagini. Negli ultimi anni, invece, sono stati indirizzati con sempre maggior successo verso problematiche delle scienze della vita.
Gli algoritmi di deep learning che sono impiegati in questo campo d’indagine hanno la capacità di apprendere analizzando un’enorme mole di dati che non potrebbero essere altrimenti processati da operatori umani.
È da questo incontro che è scaturita la soluzione al rompicapo proteico su cui gli scienziati si sono arrovellati per oltre mezzo secolo. Tali software, esaminando e confrontando le caratteristiche strutturali delle proteine già conosciute, cioè determinate per via sperimentale nel corso degli ultimi cinquant’anni, hanno imparato a generare la struttura di una nuova proteina a partire unicamente dalla conoscenza della catena di amminoacidi da cui è composta. Sarebbe stato impensabile solo pochi anni fa.
 |
Foto di Gerd Altmann via Pixabay
Mentre, infatti, è relativamente semplice conoscere la “ricetta” con cui gli organismi le preparano e la successione degli amminoacidi che le caratterizzano, le informazioni strutturali sulle proteine sono molto difficili da ricavare in laboratorio. Fino a qualche anno fa, ad esempio, potevano essere ottenute solamente le strutture delle proteine più semplici attraverso complesse procedure sperimentali e costose strumentazioni, come raggi-X, risonanze magnetiche e microscopi elettronici, spesso a disposizione solo nei principali centri di ricerca.
 |
Foto di Chokniti Khongchum via Pexels
Questi, tra l’altro, erano proprio i metodi di indagine delle strutture proteiche che conoscevo prima che mi dedicassi a giocare al Superenalotto; o meglio, questo era ciò che mi era stato consigliato in ambito accademico. In realtà non ci ho mai provato e mi sono dedicato ad altro.
Oggi, parlando di strutture tridimensionali delle proteine, ripenso a quello che una volta mi disse una persona sapendo di avere di fronte un chimico: “Mi piacerebbe guardare il mondo come lo guarda lei. Sono sicuro che vedrei molecole in tre dimensioni, come tante minuscole figure geometriche, che mi ruotano intorno in ogni momento della giornata”.
In realtà, con questi speciali occhiali “da chimico”, non si vedrebbero solamente molecole muoversi intorno a noi, ma le si osserverebbero soprattutto dentro di noi. Vi è, infatti, in ogni organismo vivente, un mondo di microscopiche strutture tridimensionali, soprattutto proteine, che hanno le più svariate forme e svolgono innumerevoli funzioni. Sarebbe un po’ come addentrarci in una città dove gli edifici siano stati progettati da architetti diversi, talvolta dai gusti decisamente eclettici, ma a partire dagli stessi materiali da costruzione.
E come per gli edifici la destinazione d’uso è spesso legata alla struttura (anche se nei tempi moderni questa non è più una regola generale), per la maggior parte delle proteine la funzione è legata alla forma tridimensionale ordinata che esse assumono nello spazio.
Da qui è facile comprendere l’importanza dei dati strutturali ottenuti con gli algoritmi di deep learning, che negli ultimi due anni hanno predetto centinaia di milioni di strutture di proteine naturali. Queste strutture sono state messe a disposizione dell’intera comunità scientifica in database liberamente accessibili online. [6-8] L’aspetto importante è che queste predizioni si sono dimostrate molto accurate e hanno superato il confronto con le più sofisticate tecniche sperimentali ampiamente validate. Tali informazioni sulle proteine naturali stanno permettendo agli scienziati in tutto il mondo di ridurre notevolmente i tempi necessari per le loro scoperte. E questo straordinario risultato ottenuto nell’ambito delle scienze della vita è testimoniato da recenti articoli apparsi su due delle riviste scientifiche più prestigiose al mondo come Science e Nature.
Occorre aggiungere che spesso la ricerca della struttura 3D delle proteine è finalizzata anche a capire l’effetto di quelle che vengono chiamate mutazioni, cioè delle modifiche nella successione degli amminoacidi rispetto alla proteina perfettamente funzionante; è in questi difetti che spesso si cela la comprensione dell’origine di una patologia. Per fare solo un esempio, è la modifica in un unico amminoacido nell’emoglobina umana a determinare quella patologia nota come anemia falciforme, caratterizzata da globuli rossi che anziché avere una normale forma a disco assumono una forma a falce. Potremmo dire che piccole differenze microscopiche sono all’origine di notevoli conseguenze a livello degli organismi.
 |
Immagine di globuli rossi di forma normale (Foto di Narupon Promvichai viaPixabay)
Provate adesso a immaginare che in una città un progettista un po’ distratto, e nei sistemi biologici si deve parlare piuttosto di casualità che di distrazione, abbia fatto realizzare una piscina olimpionica dieci centimetri più corta della lunghezza canonica di cinquanta metri. La piscina continuerà ad essere tale e forse verrà destinata ad altri nobili scopi come far divertire giovani nuotatori nel periodo estivo, o forse meno poeticamente semplicemente demolita, ma certo non avrà più la funzione per la quale era stata pensata nel progetto originario. In pratica, gli sfortunati spettatori non avranno mai occasione di vedervi gareggiare campioni olimpionici. Con le mutazioni abbiamo un problema analogo. Avremo una proteina con una diversa struttura rispetto a quella funzionante e, quindi, con una diversa funzione, cosa che spesso ha effetti deleteri sugli organismi viventi. Anche qui è facile comprendere quanto possa essere importante l’apporto dell’Intelligenza Artificiale.
Se in un primo momento l’incontro tra i due mondi, quello delle proteine e dell’Intelligenza Artificiale, ha permesso di esplorare l’universo delle molecole naturali, la nuova frontiera è addirittura quella di progettare proteine completamente nuove e non presenti in natura, aprendo un campo di indagine ancora inesplorato e potenzialmente molto fruttuoso.
Recentemente sono state mostrate le potenzialità degli algoritmi nel trovare soluzioni sempre più rapide e accurate alle sfide poste dal design di nuove proteine artificiali. [9] Queste nuove macromolecole, che permetteranno agli scienziati di esplorare zone dell’universo proteico che la natura stessa non ha ancora indagato, potranno rivelarsi potenzialmente utili in campo medico, energetico e tecnologico. Sarà come avere a disposizione un nuovo potente telescopio James Webb che nei prossimi mesi ci potrà inviare immagini di forme proteiche ancora del tutto sconosciute.
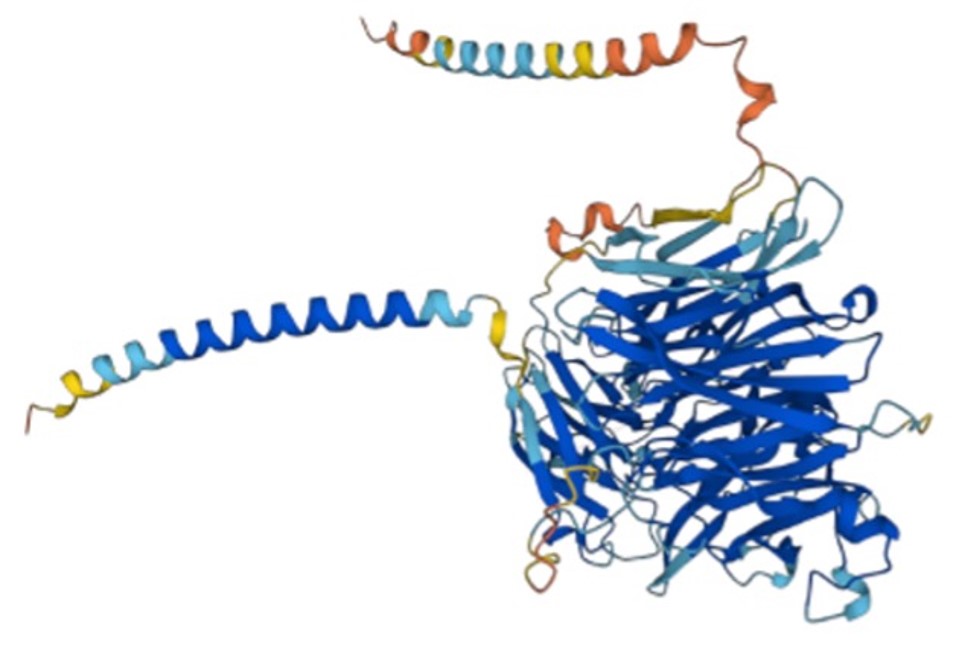 |
Modello di struttura proteica da Plasmodium falciparum (Fonte: AlphaFold Protein Structure Database, https://alphafold.ebi.ac.uk, via CC-BY-4.0 licence)
Esiste, tuttavia, un settore dell’universo delle proteine naturali che ancora in gran parte risulta inesplorato e solo parzialmente descritto dagli algoritmi di Intelligenza Artificiale: è quello rappresentato dalla galassia dalle proteine intrinsecamente disordinate. [10] Tali strutture sono state catalogate nel database DisProt, liberamente accessibile online. [11, 12]
Questa nuova galassia, scoperta da alcuni decenni, sembra governata da leggi diverse da quelle sulle quali si è basata fino ad ora la nostra tradizionale comprensione del ruolo delle proteine. Tali molecole, talvolta coinvolte anche nell’insorgenza di patologie, sono infatti in grado di svolgere le loro funzioni biologiche pur non avendo un’unica struttura tridimensionale ordinata e ben definita. Presentano, invece, un’elevata flessibilità strutturale e plasticità che le rendono capaci di modificare nel tempo la loro architettura spaziale adattandola alle diverse funzioni da svolgere. La comprensione delle regole che governano questa galassia, e della mobilità che caratterizza queste proteine, è una grande sfida posta alla comunità scientifica e all’Intelligenza Artificiale.
Quello delle proteine disordinate è, tuttavia, solo uno dei campi d’indagine in cui ancora gli algoritmi di deep learning presentano dei limiti e non riescono a sostituire gli studi sperimentali. Altre problematiche aperte riguardano la comprensione del processo di folding, cioè del modo in cui le proteine si strutturano nello spazio e degli aspetti dinamici connessi alla funzione delle macromolecole. L’immagine statica che spesso abbiamo delle proteine non rende conto, infatti, di quella che è l’elevata dinamicità che caratterizza queste molecole. [13]
Sapere se, e quando, l’Intelligenza Artificiale fornirà delle risposte anche a questi interrogativi è solo una questione di tempo.
[1] J. Jumper, R. Evans, A. Pritzel, et al., Nature, 2021, 596(7873), 583–589.
[2] J. Dauparas, I. Anishchenko, N. Bennett et al., Science, 2022, 378(6615), 49–56.
[3] E. Callaway, Nature, 2022, 608(7921), 15–16.
[4] E. Callaway, Nature, 2022, 609(7928), 661–662.
[5] E. Callaway, Nature, 2022, 611(7935), 211–212.
[6] R. F. Service, Science, 2021, 373(6554), 478.
[7] https://alphafold.ebi.ac.uk/
[9] A. Madani, B. Krause, E. R. Greene, et al., Nat. Biotechnol., 2023. DOI: 10.1038/s41587-022-01618-2.
[10] V. N. Uversky, Frontiers in Physics, 2019, 7, DOI: 10.3389/fphy.2019.00010, (https://www.frontiersin.org/articles/10.3389/fphy.2019.00010)
[11] https://disprot.org/
[12] F. Quaglia, B. Mészáros, E. Salladini, et al., Nucleic Acids Research, 2022, 50 (D1), D480–D487.
[13] G. Villani, Rappresentazioni e modelli del mondo molecolare in Immagini e strumenti digitali nella didattica delle scienze, 02/2023, Pisa University Press, 21-31.